각종 데이터 분석 실기시험 대비에 도움이 되는 파이썬 문법을 모아보았다. 그 세 번째로 통계를 다룬다.
상관분석
다양한 상관분석 기법이 있으나, 여기에서는 Pearson 상관분석과 순위 상관분석을 다룬다.
1 | ser1 = pd.Series([1, 5, 2, 7, 10, 12]) |
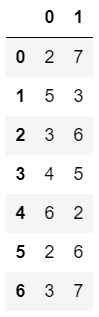
Pandas
Pandas는 DataFrame의 메서드로 .corr()을 제공한다. 별다른 옵션 없이 사용할 경우는 Pearson 상관계수를 계산한다. method 인자에는 다음과 같은 값을 입력할 수 있다.
- pearson : standard correlation coefficient
- kendall : Kendall Tau correlation coefficient
- spearman : Spearman rank correlation
1 | df_corr.corr() |
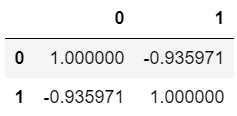
단, Pandas의 .corr() 메서드는 단순 상관계수만을 출력해주기 때문에 p-value 확인을 위해서는 SciPy 라이브러리를 활용해야 한다.
SciPy
SciPy의 함수는 상관계수 뿐만 아니라 p-value 또한 반환하기 때문에 보다 상세한 해석을 위해 사용한다. 결과는 검정통계량(왼쪽)과 p-value(오른쪽)가 tuple로 묶여서 출력된다.
1 | from scipy.stats import pearsonr |
독립성 검정
Pandas의 pd.crosstab() 으로 만든 결과물을 입력 하는 것을 추천한다.
1 | df_chi2 = pd.DataFrame({"ID": ["A", "A", "A", "A", "A", "B", "B", "B", "B", "B"], |
위 결과는 p-value가 유의수준 5%(0.05) 기준으로 해당 값 보다 크기 때문에(0.5186) 귀무가설을 기각하지 못한다.
정규성 검정
정규성 검정은 종류가 제법 많지만 대표적으로 SciPy의 shapiro() 함수로 Shapiro-Wilk 검정을 실시할 수 있다.
1 | ser_nor = pd.Series([4, 2, 5, 6, 7, 4, 5, 2, 6, 1, 3, 0, 15]) |
위 결과는 p-value가 유의수준 5%(0.05) 기준으로 해당 값 보다 작기 때문에(0.024) 귀무가설을 기각하고 대립가설을 채택한다.
등분산 검정
1 | ser_1 = pd.Series([2, 5, 3, 4, 6, 2, 3]) |
Bartlett’s test
표본이 정규성을 따를 때 사용 가능한 등분산 검정법.
1 | from scipy.stats import bartlett |
위 결과는 p-value가 유의수준 5%(0.05) 기준으로 해당 값 보다 크기 때문에(0.5499) 귀무가설을 기각하지 못한다.
Levene’s test
표본의 정규성 여부와 관계 없이 사용 가능한 등분산 검정법.
1 | from scipy.stats import levene |
위 결과는 p-value가 유의수준 5%(0.05) 기준으로 해당 값 보다 크기 때문에(0.6903) 귀무가설을 기각하지 못한다.
t-test
두 집단의 평균 차이를 검정하는 방법.
Scipy의 t-test 관련 함수는 공통적으로 결측치 처리 방법을 결정하는 nan_policy 인자가 있으나 되도록이면, 함수에 넣기 전에 결측치를 미리 처리하고 사용하는 것을 추천한다.
예제로 사용할 데이터는 다음과 같다.
1 | ser_1 = pd.Series([2, 5, 3, 4, 6, 2, 3]) |
단일 표본
SciPy의 ttest_1samp() 함수를 사용하며 표본집단을 Series로 넣고 모평균을 popmean 인자에 할당해준다.
1 | from scipy.stats import ttest_1samp |
위 결과는 p-value가 유의수준 5%(0.05) 기준으로 해당 값 보다 작기 때문에(0.0465) 귀무가설을 기각하고 대립가설을 채택할 수 있다.
대응 표본
SciPy의 ttest_rel() 함수를 사용하며 각 표본집단을 Series로 넣어준다.
1 | from scipy.stats import ttest_rel |
위 결과는 p-value가 유의수준 5%(0.05) 기준으로 해당 값 보다 크기 때문에(0.2683) 귀무가설을 기각하지 못한다.
독립 표본
SciPy의 ttest_ind() 함수를 사용하며 각 표본집단을 Series로 넣어준다.
독립 2표본 t-test는 등분산 가정에 따라 equal_var 인자의 값을 다르게 준다.
1 | from scipy.stats import ttest_ind |
위 코드에서 1번 코드는 두 표본의 등분산, 2번 코드는 이분산으로 설정하고 계산하였다.
두 결과 모두 다 p-value가 유의수준 5%(0.05) 기준으로 해당 값 보다 크기 때문에 귀무가설을 기각하지 못한다.
One-way ANOVA
일원 분산분석은 SciPy와 StatsModels 라이브러리에서 각각 지원하며, 상세한 결과가 필요한 경우 StatsModels 라이브러리의 함수를 사용하는 것을 권장한다.
예제로 사용할 데이터는 다음과 같다.
1 | ser_1 = pd.Series([2, 5, 3, 4, 6, 2, 3]) |
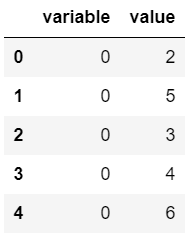
SciPy
- f_oneway(): Series 객체를 그룹별로 나눠서 입력
※ f_oneway(df[“group1”], df[“group2”], df[“group3”])
1 | from scipy.stats import f_oneway |
위 결과는 p-value가 유의수준 5%(0.05) 기준으로 해당 값 보다 크기 때문에(0.0884) 귀무가설을 기각하지 못한다.
StatsModels
StatsModels의 ols() 함수와 anova_lm() 함수를 불러와야 하기 때문에 SciPy의 함수보다 쓰기 번거롭다.
1 | from statsmodels.formula.api import ols |
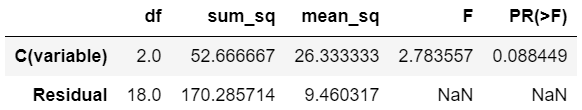
위 결과는 p-value가 유의수준 5%(0.05) 기준으로 해당 값 보다 크기 때문에(0.0884) 귀무가설을 기각하지 못한다.
사후 검정
일원 분산분석에서 귀무가설을 기각하고 대립가설을 채택했을 때 어떤 집단간 평균이 유의미하게 차이가 나는지 확인하는 방법. 대표적으로 Tukey’s HSD가 있다.
1 | from statsmodels.stats.multicomp import pairwise_tukeyhsd |
위 결과의 경우 reject는 두 집단의 평균이 같다는 귀무가설을 기각하는지 여부를 알려준다. 즉, 모든 그룹간 평균이 유의미하게 다르다고 하기 어렵다(False).
<실기시험 대비 시리즈 - Python>
Python 실기시험 대비 정리 노트 - 00
Python 실기시험 대비 정리 노트 - 01
Python 실기시험 대비 정리 노트 - 02
Python 실기시험 대비 정리 노트 - 03
Python 실기시험 대비 정리 노트 - 04


