여러 코로나 바이러스 정보 제공 웹사이트 중에서 국제적으로 유명한 사이트가 몇 개 있지만 그 중 하나인 Johns Hopkins의 코로나 바이러스 현황 대시보드를 소개하고자 한다.
존스 홉킨스는 특히나 의학대학원이 유명한데 존스 홉킨스 대학교의 명성은 의대에서 다 끌어올리는 것 같다. 관련해서는 위키피디아 및 기타 사이트를 참조하자.
아무튼 존스 홉킨스 공대에서 ArcGIS를 활용한 공개 대시보드를 만들었다.

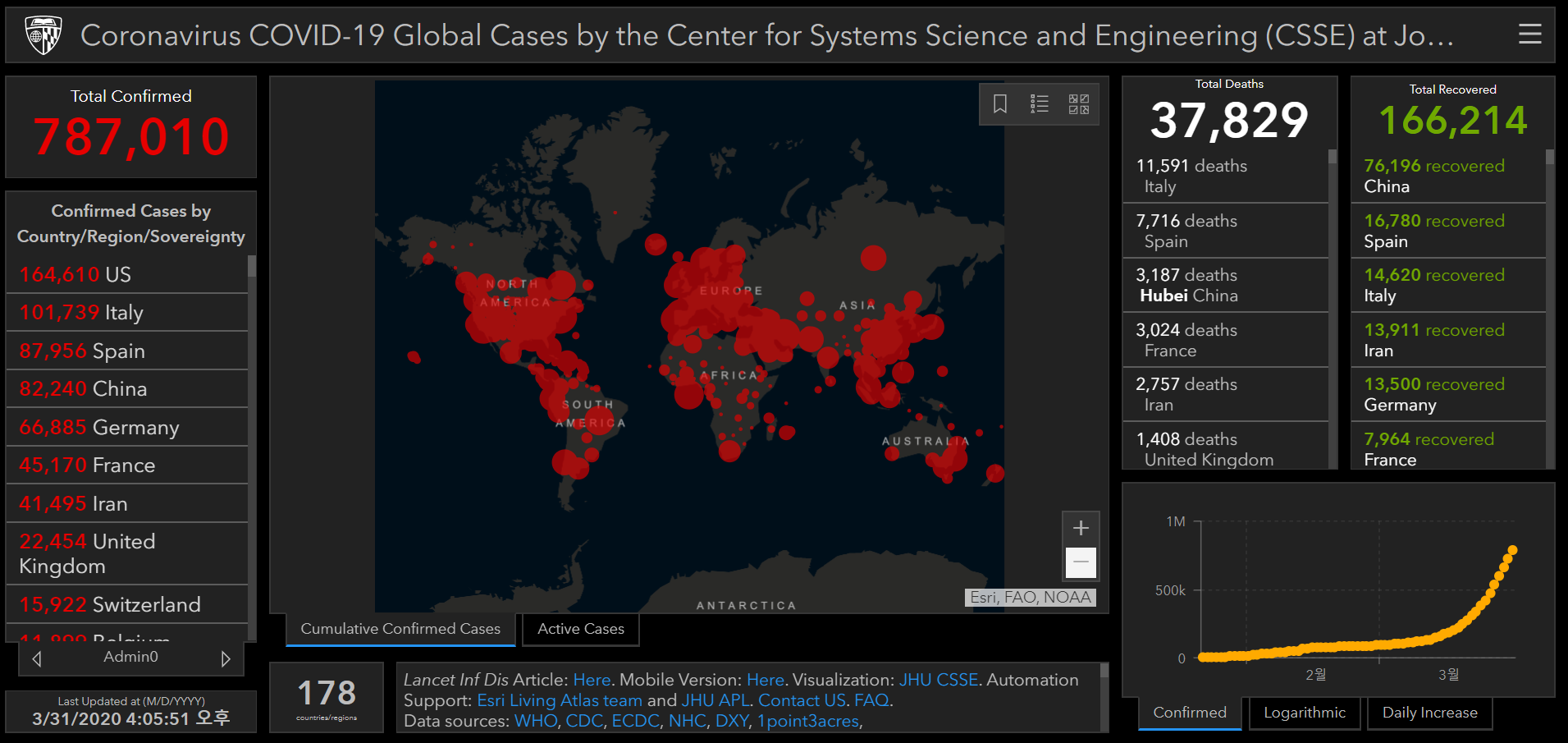
24시간 내내 해당 대시보드를 최신화 해주는 것 같은데 도대체 데이터는 어디에 있을까? 다행이도 깃헙(github)에 일별로 데이터를 업데이트 해주고 있다.
물론 깃헙을 능숙하게 사용하는 사람이라면 fork 이후에 pull을 하겠지만 그렇지 못한 사람은 데이터가 있는 페이지에서 손으로 직접 다운아야 할 것이다.
다행이도 해당 저장소(repository)는 공개 저장소라 크롤링으로 수집이 가능하다.
최초 코드는 다음과 같다.
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
|
library("rvest")
source = read_html("https://github.com/CSSEGISandData/COVID-19/tree/master/csse_covid_19_data/csse_covid_19_daily_reports")
source %>%
html_nodes(css = "tbody a") %>%
html_attr("href") %>%
grep(pattern = "csv$", value = TRUE) %>%
gsub(pattern = "blob/", replacement = "")-> file_uri
file_url = paste0("https://raw.githubusercontent.com", file_uri)
df = data.frame()
for(file in file_url){
df_sub = read.csv(file, encoding = "UTF-8")
df = rbind(df, df_sub)
}
|
위 코드는 데이터를 읽고 데이터의 주소 추출 및 정제하여 최종적으로 반복문을 통해 파일을 합치는 코드이다.
하지만 여기서 문제가 발생했다. (30분 만에 포스팅 끝내나 했는데…) 2월 1일 데이터의 경우 첫 번째 변수명이 다음과 같이 변경되어있다.
X.U.FEFF.Province.State Province.State
그래서 read.csv() 함수를 다음과 같이 수정하여 돌려보았다.
1
2
| df_sub = read.csv(file, encoding = "UTF-8",
header = FALSE, skip = 1)
|
또 문제가 발생했다. 3월 1일 부터는 각 지역의 위경도(Latitude: 위도, Longitude: 경도) 좌표가 추가되어있어 변수 개수가 맞지 않아 rbind() 함수의 동작에서 에러가 발생했던 것이다.
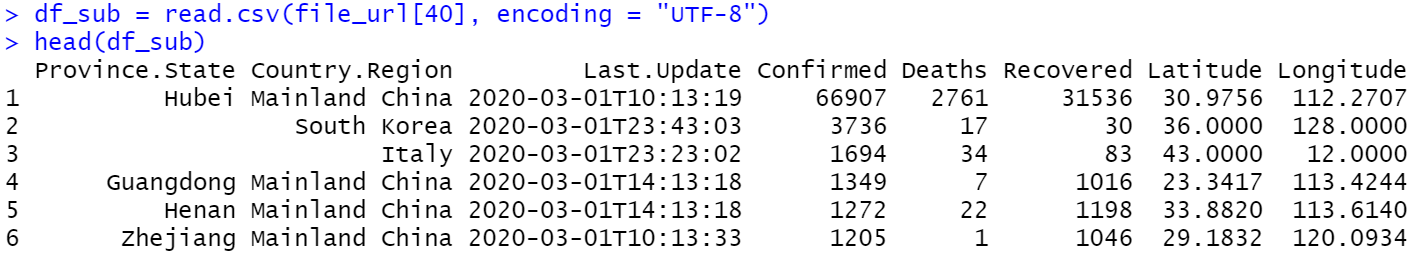
일단 위경도 정보는 제거하기로 하고 data.table 패키지의 fread() 함수를 사용하기로 했다. fread() 함수의 select 인자를 활용하면 원하는 위치 또는 이름의 변수를 가져올 수 있다. 수정한 코드는 다음과 같다.
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
|
library("rvest")
library("data.table")
source = read_html("https://github.com/CSSEGISandData/COVID-19/tree/master/csse_covid_19_data/csse_covid_19_daily_reports")
source %>%
html_nodes(css = "tbody a") %>%
html_attr("href") %>%
grep(pattern = "csv$", value = TRUE) %>%
gsub(pattern = "blob/", replacement = "")-> file_uri
file_url = paste0("https://raw.githubusercontent.com", file_uri)
df = data.frame()
for(file in file_url){
df_sub = fread(file, encoding = "UTF-8", header = FALSE,
data.table = FALSE, select = 1:6)
df = rbind(df, df_sub)
}
|
에러 없이 무사히 코드가 돌아갔다. 그래서 데이터를 조금 가공해보려고 하는데 데이터 상태가 이상했다. 어디서 발생한 문제인지 확인을 해 본 결과 3월 22일 자료 부터 다시 한 번 데이터 형식이 바뀌었다.
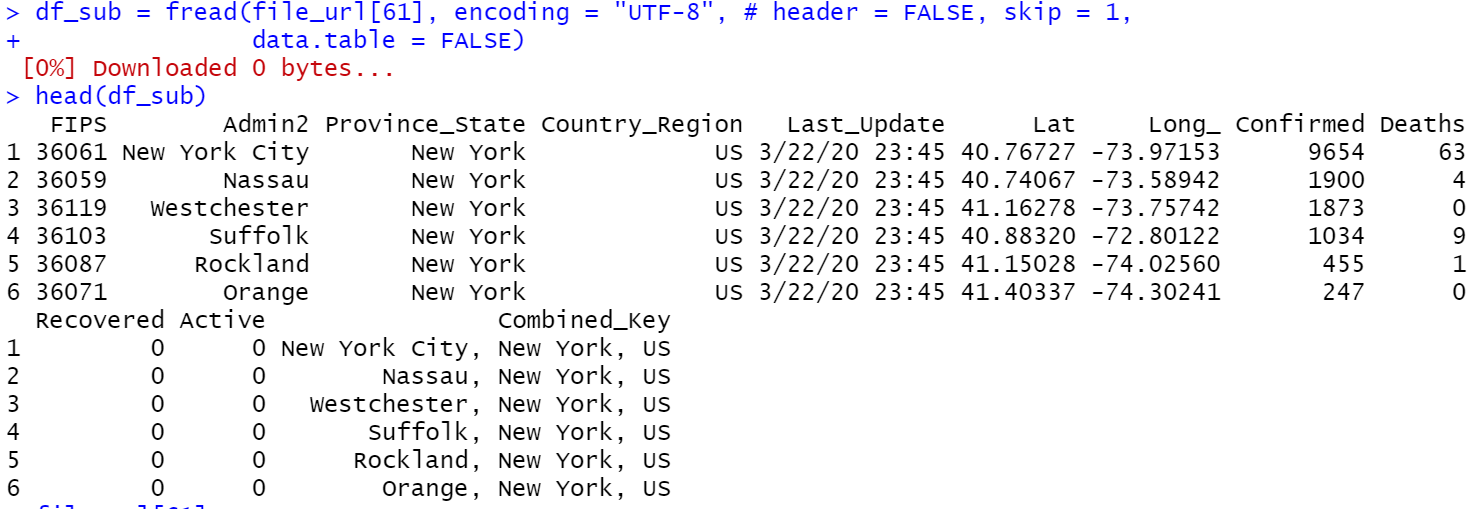
이것 까지 보니 딱 하기 싫어졌다. 계속 데이터를 크롤링으로 수집하게 되면 ip가 막힌다던지 정보 제공자에게 피해가 갈 수 있기 때문에 데이터를 각각 다운받는 코드로 바꾼 코드를 공유하고 마치도록 하겠다.
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
| library("rvest")
source = read_html("https://github.com/CSSEGISandData/COVID-19/tree/master/csse_covid_19_data/csse_covid_19_daily_reports")
source %>%
html_nodes(css = "tbody a") %>%
html_attr("href") %>%
grep(pattern = "csv$", value = TRUE) %>%
gsub(pattern = "blob/", replacement = "")-> file_uri
file_url = paste0("https://raw.githubusercontent.com", file_uri)
file_path = gsub(pattern = "^.*?reports/", replacement = "", file_url)
for(n in 1:length(file_url)){
download.file(url = file_url[n],
destfile = file_path[n],
mode = "wb")
}
|
해당 데이터를 전처리하는 포스트는 짜증이 가라 앉은 이후 가까운 미래에 작성할 수 있도록 하겠다.






